Ngày cập nhật 13 Tháng Mười Hai 2019
Sàng lọc đột biến gen trong đậu nành [Glycine max(L.) Merr.] đã bị giới hạn bởi bộ gen của chúng. CRISPR ‐ Cas9 có thể tạo đột biến đa bội trong các cây trồng có bộ gen phức tạp. Tuy nhiên, hiệu quả chuyển đổi của đậu nành vẫn còn thấp và vẫn là trở ngại lớn trong việc áp dụng CRISPR ‐ Cas9 như một công cụ sàng lọc đột biến.
Một vector CRISPR ‐ Cas9 mới, pGES201, đã được phát triển để chỉnh sửa bộ gen trong đậu nành. Trong vectơ này, BASTA đã được sử dụng làm điểm đánh dấu lựa chọn chuyển đổi và mã hóa đậu nành sp spCas9 được tối ưu hóa được điều khiển bởi một promoter nội sinh mạnh, proGmSCREAM M4 ( pM4 ).
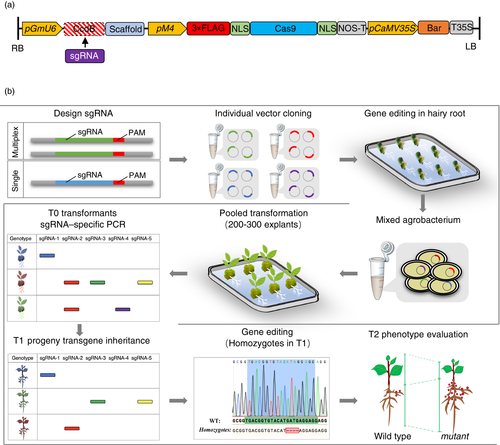
Hình ảnh: Một quy trình gây đột biến CRISPR ‐Cas9 gộp trong đậu nành. (a) sgRNA được thiết kế để nhắm mục tiêu nhiều locus hoặc một locus cụ thể và các vectơ CRISPR được nhân bản riêng lẻ. Trong quy trình tối ưu hóa, hiệu quả chỉnh sửa gen của từng sgRNA được đánh giá trong rễ tơ. Các vectơ hiệu quả cao được gộp vào một thư viện con trong A. tumefaciens, sau đó được chuyển vào đậu nành. Các cây T0 và T1 được tạo kiểu gen bởi SSP (các thanh màu cho biết sgRNA dương tính) và chỉnh sửa gen được xác minh bằng trình tự PCR và Sanger. Các đột biến đồng hợp tử mong muốn sẽ thu được ở thế hệ T1 hoặc T2 và sau đó phải chịu kiểu hình. (b) Cấu trúc của vectơ pGES 201. Các kiểu hạn chế IIS BsaI đã được sử dụng để nhân bản sgRNA giữa promoter U6 và sgRNA. Ở giữa hai vị trí hạn chế BsaI là một gen độc tố, mp4B. pM4 đã được sử dụng điều khiển spCas9. BASTA điều khiển 35S là chỉ thị được lựa chọn.
Nghiên cứu trên nền tảng CRISPR ‐ Cas9 được gộp chung để tạo ra các quần thể đột biến gen đậu tương đậu nành. Nghiên cứu đã tối ưu hóa các bước chính trong giao thức sàng lọc bao gồm xây dựng véc tơ, đánh giá sgRNA, chuyển đổi gộp, xác định sgRNA và xác minh chỉnh sửa gen và đã xây dựng 70 vectơ CRISPR ‐ Cas9 để nhắm mục tiêu 102 gen ứng tiềm năng và parolog của chúng đã bị biến đổi gộp thành 16 khối. Một quần thể bao gồm 407 dòng T0 đã thu được chứa tất cả các sgRNA với tần số đột biến trung bình là 59,2%, bao gồm 35,6% dòng mang đột biến đa bội. Tần số đột biến trong thế hệ con cháu T1 có thể tăng hơn nữa mặc dù có được một thể khảm biến đổi gen. Trong quần thể này, nghiên cứu đặc trưng đột biến kép gmric1/gmric2 với số lượng nốt tăng và gmrdn1‐1 / 1‐2 / 1‐3 ba dòng đột biến với số nốt sần giảm. Nghiên cứu của các tác giả cung cấp một chiến lược tiên tiến để tạo ra một quần thể đột biến gen đa mục tiêu để khắc phục vấn đề dư thừa gen ở đậu nành cũng như trong các cây trồng chính khác.
Nguồn: onlinelibrary.wiley.com